NG | 华中农大棉花团队构建陆地棉Jin668-T2T基因组揭示棉花体细胞胚胎发生分子机制赋能精准基因编辑

研究背景:
棉花作为全球最重要的经济作物之一,广泛应用于纺织、油脂、饲料等多个领域。在满足人类基本生活需求的同时,棉花育种也面临诸多挑战,其中“基因型依赖的遗传转化难题”尤为突出。虽然CRISPR/Cas等基因编辑技术为作物遗传改良带来了革命性的契机,但其成功实施高度依赖于受体材料的转化与再生能力。
在现有体系中,陆地棉的转化与再生普遍依赖于极少数高再生基因型,如美国珂字棉系列、泗棉3号、中棉所24、YZ1、Jin668等,其中YZ1和Jin668为华中农业大学棉花团队经过20余年通过大规模基因型筛选和连续再生驯化策略培育的两个具高再生能力的陆地棉基因型材料,其中Jin668已经被全球100多个课题组引进,成为全球棉花遗传转化和基因编辑最主要的受体材料。而这些材料虽然有较好的再生能力,但因为选育年代久远,农艺性状难以满足当代棉花育种的需求,而其他绝大多数农艺优良品种表现为“难转化、难再生”。这种严重的基因型依赖性,不仅限制了棉花功能基因组的研究,也严重制约了棉花分子设计育种的效率。尤其是在当前“精准育种”和“智能化设计”成为主流趋势的背景下,开发更加高效、普适的转化再生平台显得尤为迫切。基于顶端分生组织的“非基因型依赖”转化系统扩大了转化品种范围,但仍普遍面临转化效率低、嵌合率高以及基因编辑效率低等问题。而近些年在禾本科植物如玉米、小麦中通过精准调控再生相关因子(BBM、Wushel、GRF/GIF 等),有效地的破解植物遗传转化基因型依赖的瓶颈。因此,解析Jin668等优良基因型的高效再生机制,将为非基因型依赖的植物遗传转化体系的推广应用提供关键的靶标和重要理论支撑。
研究内容与成果:
1、精准解析 Jin668和YZ-1 基因组
为实现完整的基因组组装,本研究以Jin668 自交系为研究对象,综合利用多种测序平台进行深度测序。其中,基于 Nanopore 技术获得的超长读长数据总量达 387.02 Gb(约 169×,N50 = 52.26 Kb,包括约30×的超长片段 >100 Kb),PacBio HiFi 高精度测序获得 124.88 Gb 数据(覆盖度约为54×)。初步组装结果高度连续,仅由 44 个contig组成,其中17条 contig 可直接对应染色体。未能解决的9个片段区主要集中在高度重复、超长串联重复区域。在初步组装基础上,进一步利用Hi-C数据进行组装校正与染色体构建,将剩余23个contig锚定至9条染色体上,但仍存在14个组装缺口。随后,联合使用ONT、HiFi、Illumina短读长及先前HiFi版本的草图序列,开展缺口填补、端粒补齐与重复序列校正,最终获得完整的 Jin668 T2T 基因组(图1)。利用端粒重复序列 AAACCCT/AGGGTTT进行全基因组端粒定位,共在26条染色体两端识别出全部52个端粒序列,并通过FISH实验验证了其物理定位。此外,还结合FISH实验验证了位于A09、D07和D09染色体上的约8.1 Mb的5S rDNA和 75.1 Mb的主45S rDNA重复区(图2),进一步增强了组装可信度。
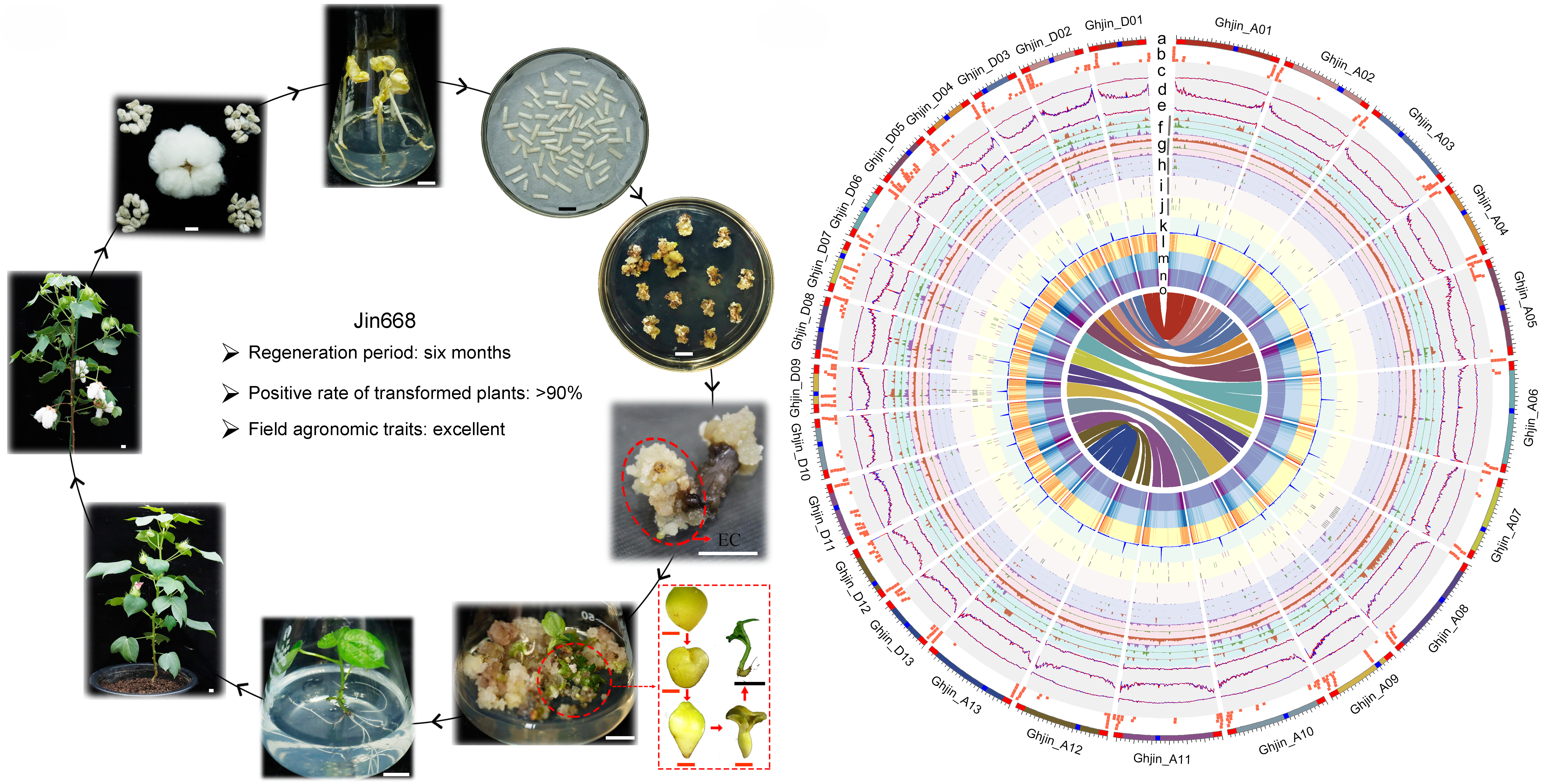
图1. Jin668组培过程和T2T基因组。
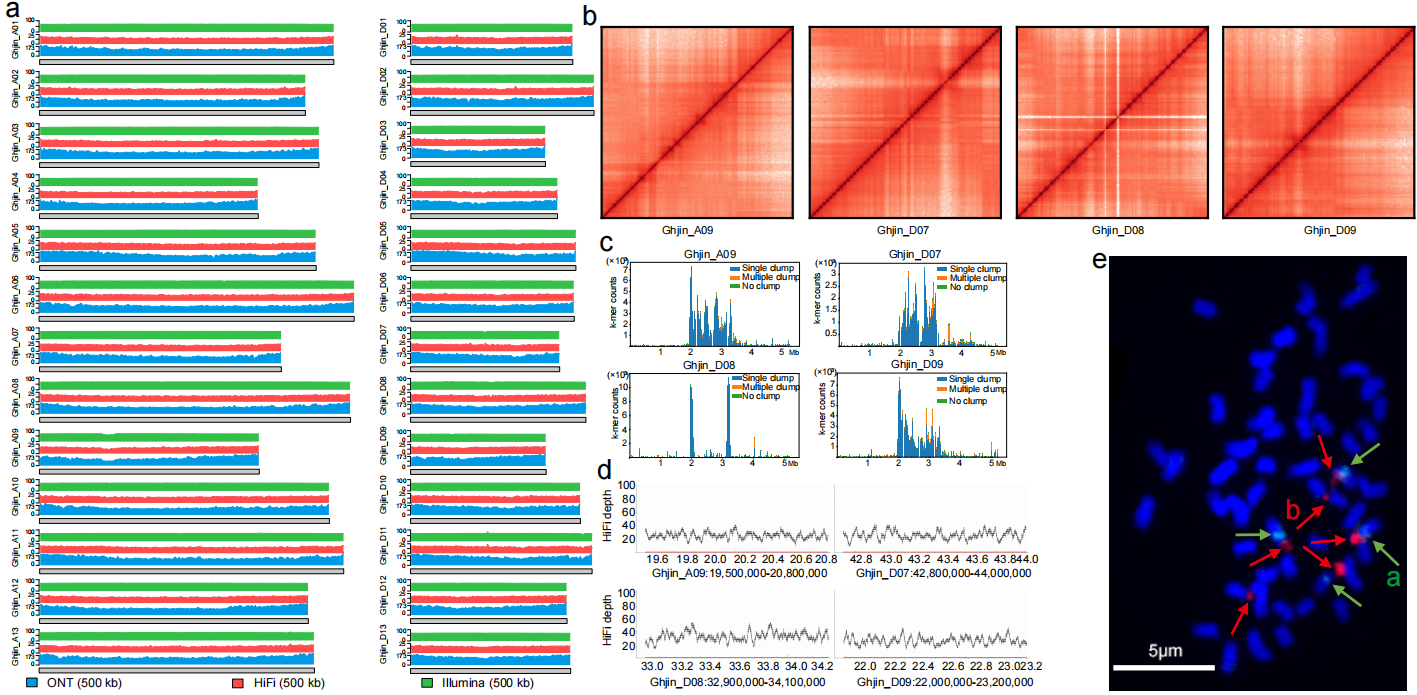
图2. 基于比对和FISH实验的组装验证。
2、揭示棉花染色体着丝粒的复杂结构与演化特征
借助Jin668的T2T基因组,通过Jin668特异的CENH3染色质免疫共沉淀实验和高质量测序,鉴定了每条染色体上1.0~1.8 Mb的着丝粒核心区域,总长度大约30.1 Mb。进一步对着丝粒区域的转座子序列进行鉴定和注释,发现Jin668的着丝粒区域主要被CRM与Tekay两类反转录转座子入侵构成。此外,还在Ghjin_D08染色体上,发现了包含大约3543个拷贝的194 bp重复单元组成的高阶重复结构(HOR),并通过FISH实验确认其精准定位于着丝粒原点。而且,除在着丝粒区域形成HOR外,该重复单元还在近着丝粒区域(pericentromeric region)串联重复出现,其总长度约为682 Kb,且与着丝粒区域的HOR间隔约4 Mb(图3)。此外,研究还发现着丝粒区的GC含量、甲基化水平及组蛋白修饰特征(如H3K4me3和H3K27ac)与其功能状态密切相关。这一系统性研究描绘出棉花染色体核心区域的“结构地图”,为理解其遗传稳定性和基因表达调控提供重要线索。
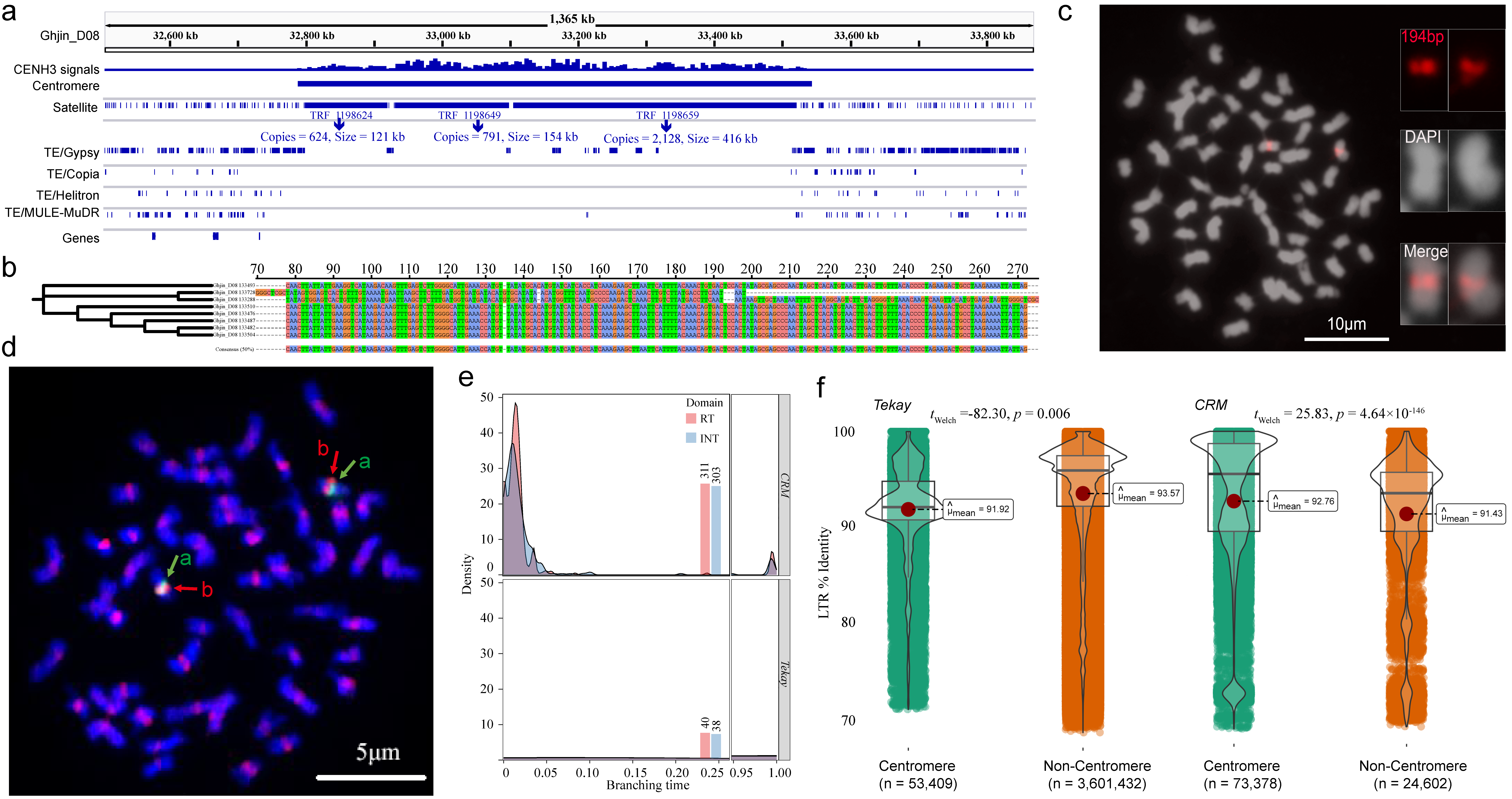
图3.Jin668的着丝粒特征。
3、基因组对比揭示高再生基因型的遗传特征
通过比较高再生能力基因型(Jin668、YZ1、ZM24)与不可再生基因型(TM-1),研究揭示了控制愈伤组织形成与胚性转化的潜在基因组基础。三大再生基因型共同特有约560个基因,富集于激素调控相关过程,如极性转运、细胞分裂、细胞命运决定等。此外,研究还系统比对四个基因组,识别出超过100万个SNP、24万个InDel、数十处结构变异和560个PAVs,并发现这些变异与已知SE相关基因呈显著关联。进一步分析发现,TE插入在TM-1中更频繁地出现在SE基因启动子区,尤其是Mutator类转座子。代表性基因如LEC、WOX4及AUX1等,其TE插入状态影响表达模式,从而可能调控再生潜力(图4)。该结果为理解棉花再生分化能力的遗传基础和分子机制提供了关键视角。
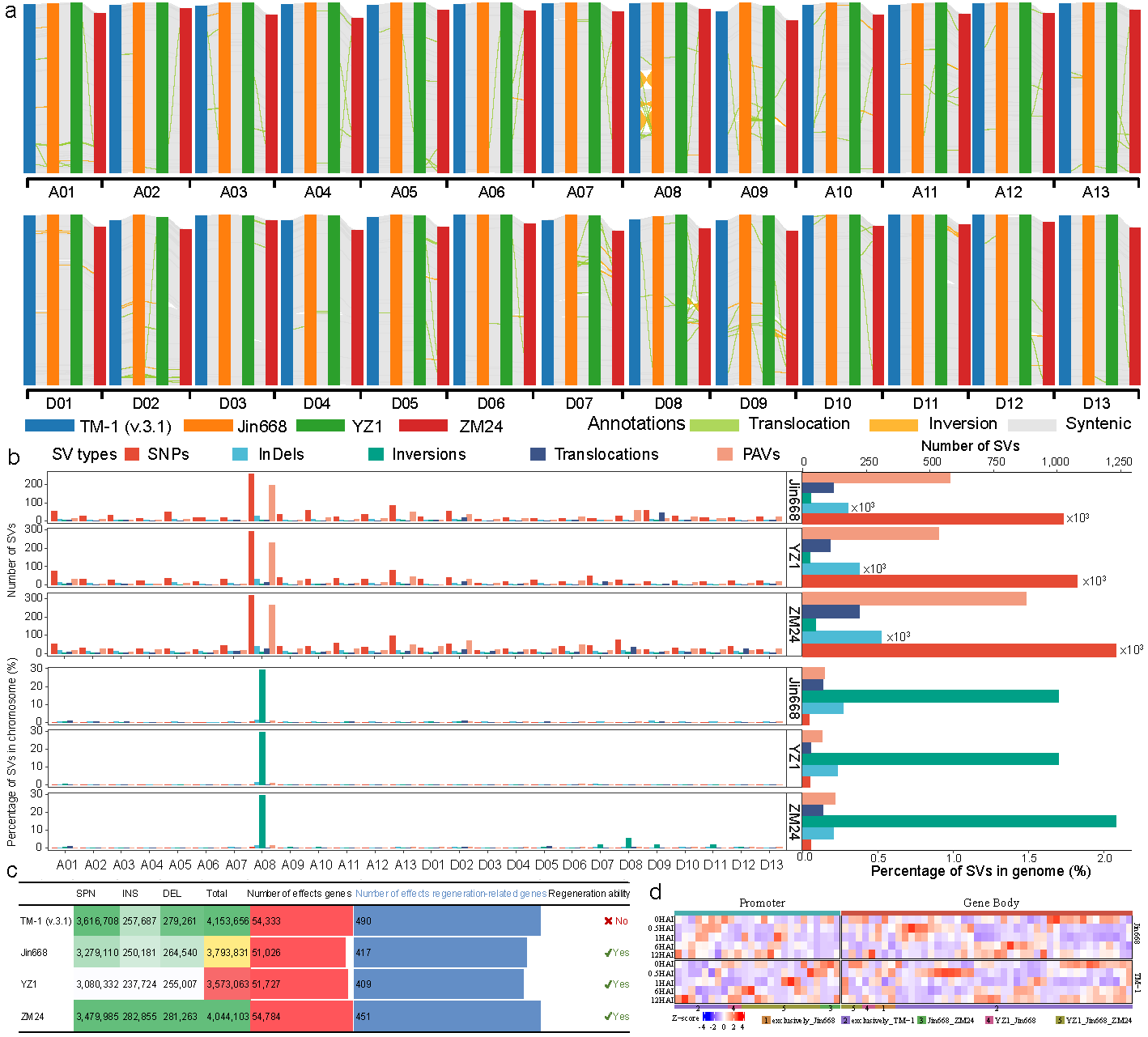
图4. 可再生基因型Jin668、YZ1和ZM24与不可再生基因型TM-1的比较分析。
4、表观调控与转录动态描绘棉花初始再生过程
采用ATAC-seq和RNA-seq技术,对Jin668与TM-1在愈伤组织诱导初期(0–12小时)进行了系统采样,解析其染色质可及性与基因表达变化。在再生基因型中观察到逐渐增强的染色质开放性,伴随关键转录因子(如AGL15、WOX、LEC2、ERF等)表达上调。而TM-1中则呈现开放性下降与基因沉默。我们进一步构建转录因子结合位点数据库,揭示Jin668在各阶段特异富集调控胚性发育与激素极性的TFs。此外,重建的SE共表达网络显示,调控愈伤形成、乙烯响应、激素信号与表观修饰的核心基因(如PIN、SAUR、MYC2、DdRP等)密切协同,形成模块化调控体系(图5)。该研究为棉花再生启动阶段的分子调控图谱提供了前所未有的动态视角,也为再生效率提升提供理论支持。
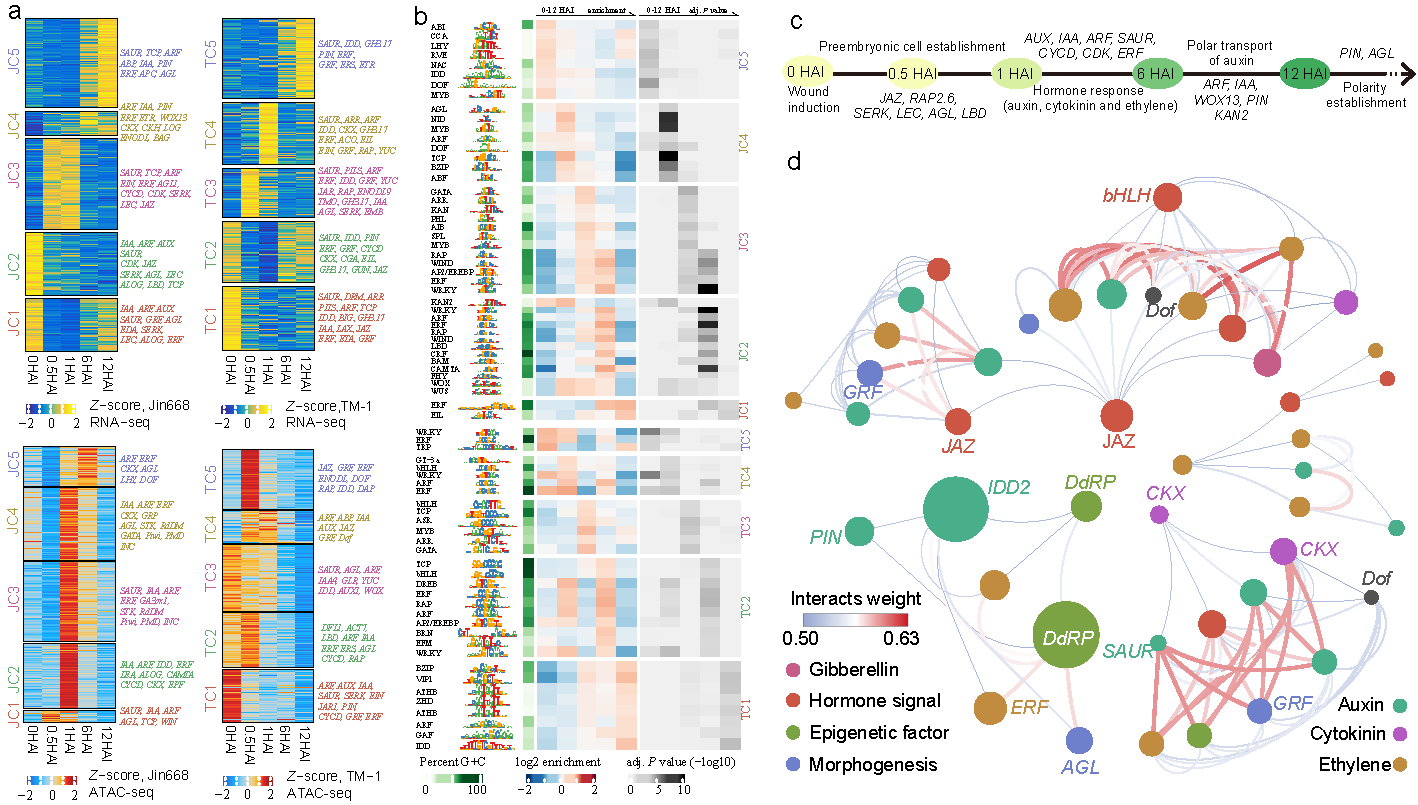
图5. 体细胞胚胎发生过程中Jin668和TM-1的染色质可及性和基因表达的动态变化
5、AGL15 是棉花SE再生的关键调控因子
为验证关键基因在SE再生中的功能,研究聚焦于AGL15与AUX1,并构建CRISPR敲除与过表达载体,在Jin668、YZ1和TM-1中进行遗传转化实验。结果显示,AGL15缺失显著降低愈伤组织增殖速率(CPR)和胚性分化能力,而其过表达则能显著提升YZ1和TM-1的再生能力,尤其在60天时效果显著。相比之下,AUX1的过表达未能有效改善TM-1再生性,表明其对SE的促进作用依赖于特定遗传背景。切片分析进一步验证了AGL15在维管组织细胞增殖中的正调控作用(图6)。该发现不仅首次从功能层面验证了AGL15在棉花再生中的核心作用,也为后续优化转化体系提供了靶点。
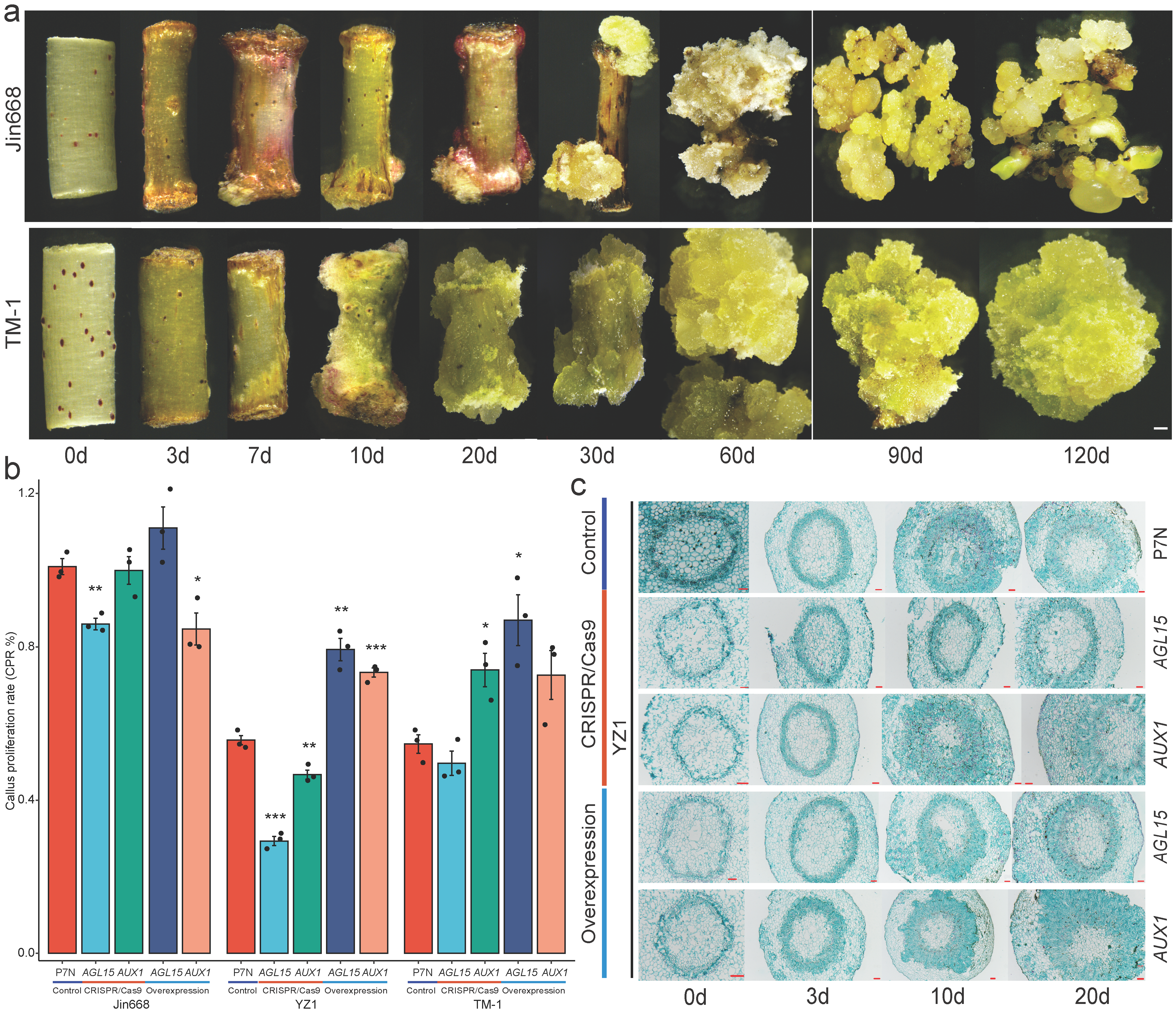
图6. 再生相关基因的功能验证。
6、Jin668基因组助力精准基因编辑设计
由于过去缺乏Jin668参考基因组,CRISPR编辑多依赖TM-1参考序列,存在显著脱靶风险。研究系统评估了TM-1、Jin668和ZM24间的基因差异对Cas9与Cas12a靶位设计的影响,发现不同基因组间的PAM识别位点及高质量sgRNA数量存在明显波动。在分析1:1:1同源基因中,Jin668参考可识别更多精准靶点,并发现大量TM-1中遗漏的脱靶位点(图7)。进一步转化实验证实:仅在Jin668中观察到有效编辑事件,而TM-1未检出突变。这表明基因组差异显著影响编辑效率(图8)。
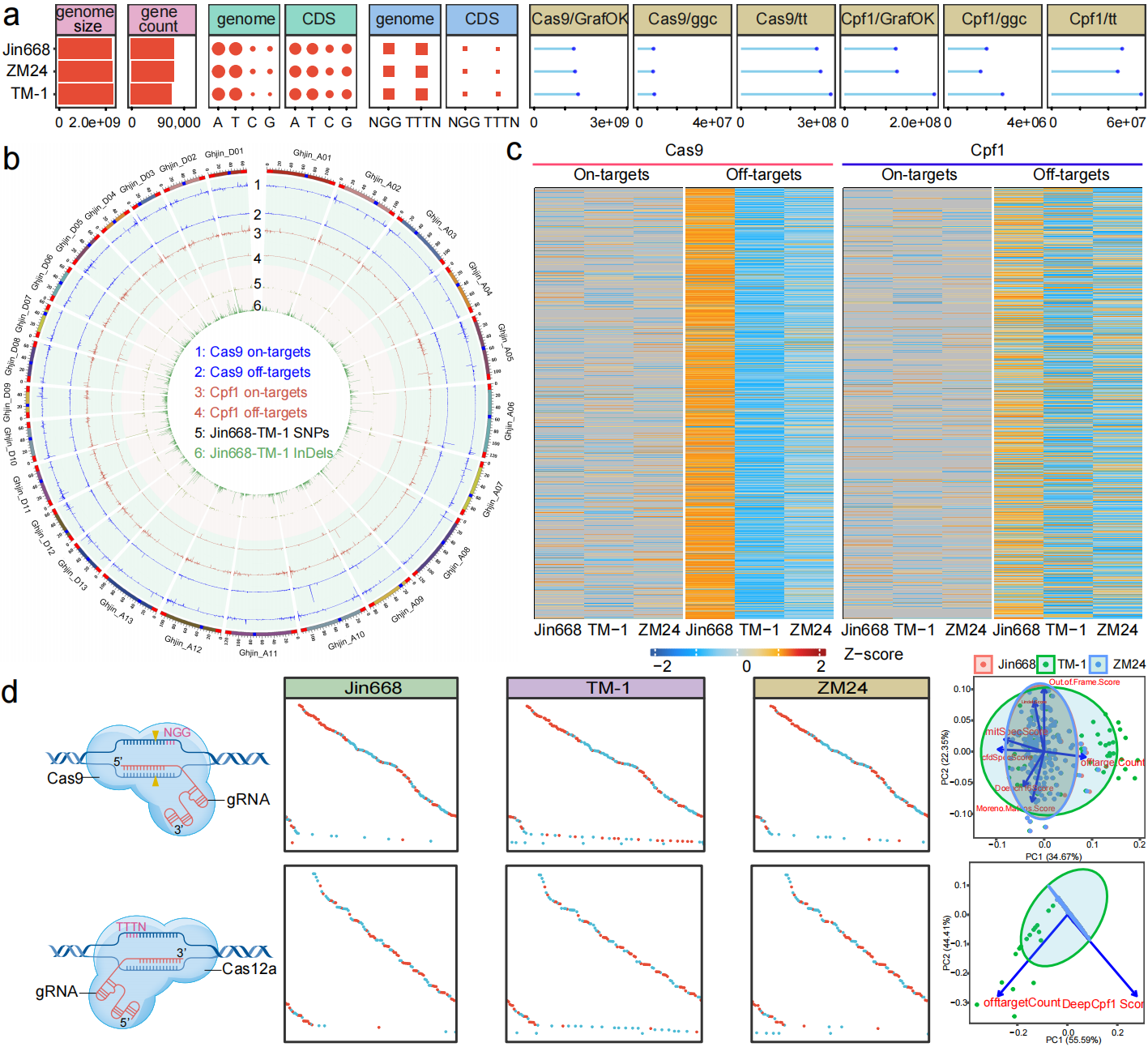
图7. 棉花遗传变异极大地影响了基于CRISPR的基因组编辑的准确性。
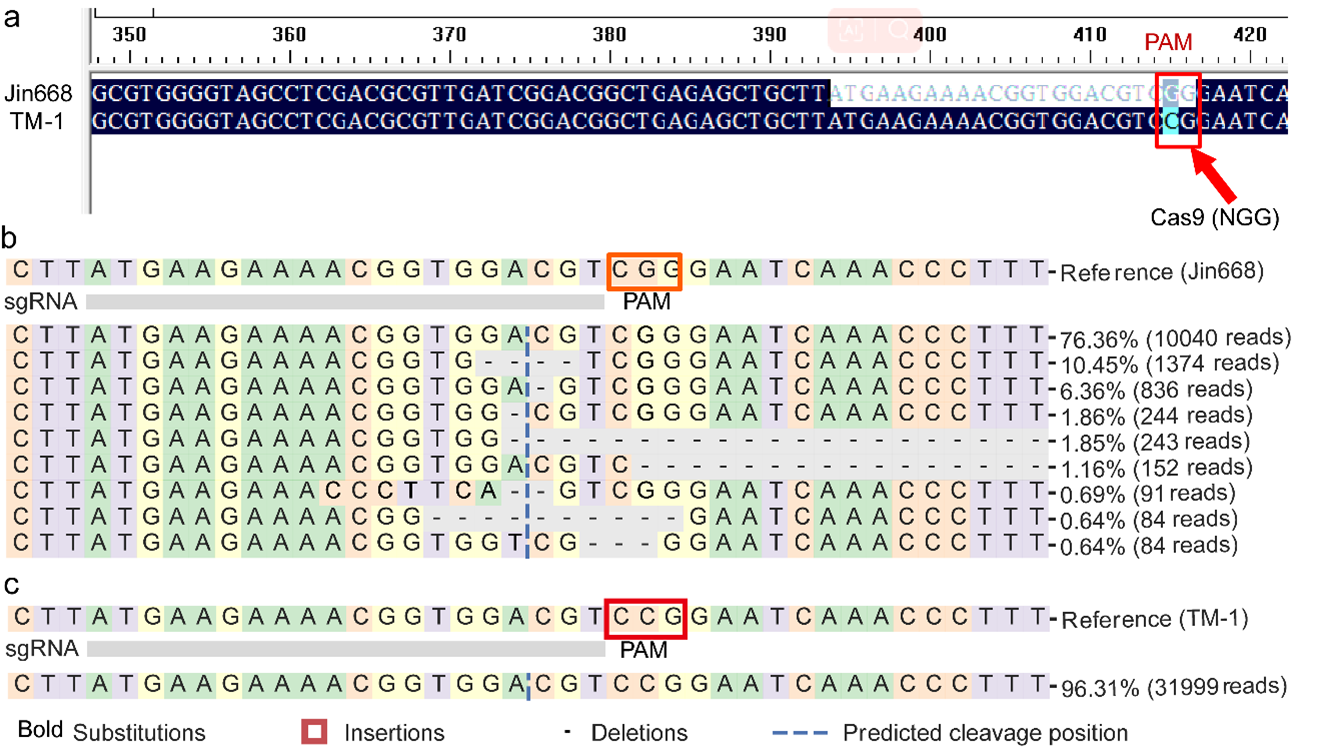
图8. 遗传转化实验表明,Jin668基因组提高了sgRNA设计的准确性。
图9. T2TCotton-Hub基因组数据库。
研究亮点:
-
构建陆地棉高再生基因型Jin668的端到端(T2T)完整参考基因组和YZ1的基因组近完成图(鉴定到45个端粒,整个基因组仅存在72个缺口);
-
精准解析了Jin668-T2T基因组大约30.1 Mb着丝粒区结构,发现位于着丝粒区的CRM/Tekay类反转座子和D08染色体特异的由194 bp重复单元构成的高阶重复区域;
-
系统鉴定出Jin668-T2T基因组大约75.1 Mb的45S rDNA簇和大约8.1 Mb的5S rDNA簇,并通过FISH实验进行证实;
-
揭示高再生能力基因型体细胞再生的遗传基础和调控网络,确定AGL15为关键因子,其过表达显著提升再生效率;
-
系统评估并证实基因型间的遗传变异显著影响基于CRISPR的基因组编辑的sgRNA识别位点,T2T-Jin668参考基因组显著提升CRISPR编辑精度;
-
基于T2T-Jin668参考基因组和本研究数据集建立T2TCotton-Hub在线平台(http://jinlab.hzau.edu.cn/T2TCottonHub/),集成多组学资源,助力棉花功能基因研究与精准育种。
研究意义:
本研究以高再生基因型Jin668为基础,构建了棉花端粒到端粒(T2T)级别的高质量参考基因组,并结合转录组、ATAC-seq、功能验证等手段,系统揭示了再生能力的调控网络与CRISPR系统靶点优化机制。研究不仅为棉花构建高效、通用的遗传转化平台奠定了理论基础,也为棉花功能基因组的研究提供了重要的资源。未来,T2T基因组将在多倍体进化、表观调控、结构变异解析等方面展现更大潜力,同时也将成为推动AI辅助靶点设计与大规模基因组编辑应用的关键支点,加速实现高效、精准、智能的作物设计育种新时代。
