NC | 华中农业大学棉花遗传改良团队发布海岛棉高质量图泛基因组:解析纤维品质及产量遗传基础助力优质棉育种
近日,华中农业大学作物遗传改良全国重点实验室张献龙院士团队联合国内外多家科研机构,在国际权威期刊《Nature Communications》上发表了题为“Pangenome analysis reveals yield- and fiber-related diversity and interspecific gene flow in Gossypium barbadense L.”的研究论文。该研究通过从头组装12个野生-驯化连续体的海岛棉基因组,构建了首个海岛棉高质量泛基因组,并解析了其纤维品质和产量相关性状的遗传变异及种间渐渗现象。研究成果鉴定出棉花纤维产量与品质遗传改良的关键基因位点,提供了丰富的遗传资源,为海岛棉同步提升产量与纤维品质提供了新思路。

海岛棉(Gossypium barbadense L.)拥有世界顶级的纤维品质,其纤维以长度长、韧性强、光泽度高、垂感好等特点著称,是纺制精、细、薄高端棉纺织品的必备原料。然而,海岛棉适应性差且产量低,导致市场长期供不应求,我国优质棉长期依赖进口。为破解这一瓶颈问题,团队前期已构建了海岛棉基因组草图(Yuan et al., 2015)和参考基因组(Wang et al., 2019),并解析了其变异图谱(Yuan et al., 2021)。为进一步探究海岛棉的遗传多样性并鉴定其优异纤维品质的遗传位点,本研究在前期构建的四倍体棉花系统发生树(Yuan et al., 2021)基础上,筛选出12份涵盖野生-驯化连续体的海岛棉种质资源,包括:7份源自起源地附近的原始种质、2份来自加勒比海地区的农家种及3份广泛种植的栽培种。利用HiFi测序技术,研究团队完成了这12份海岛棉高质量基因组的从头组装。
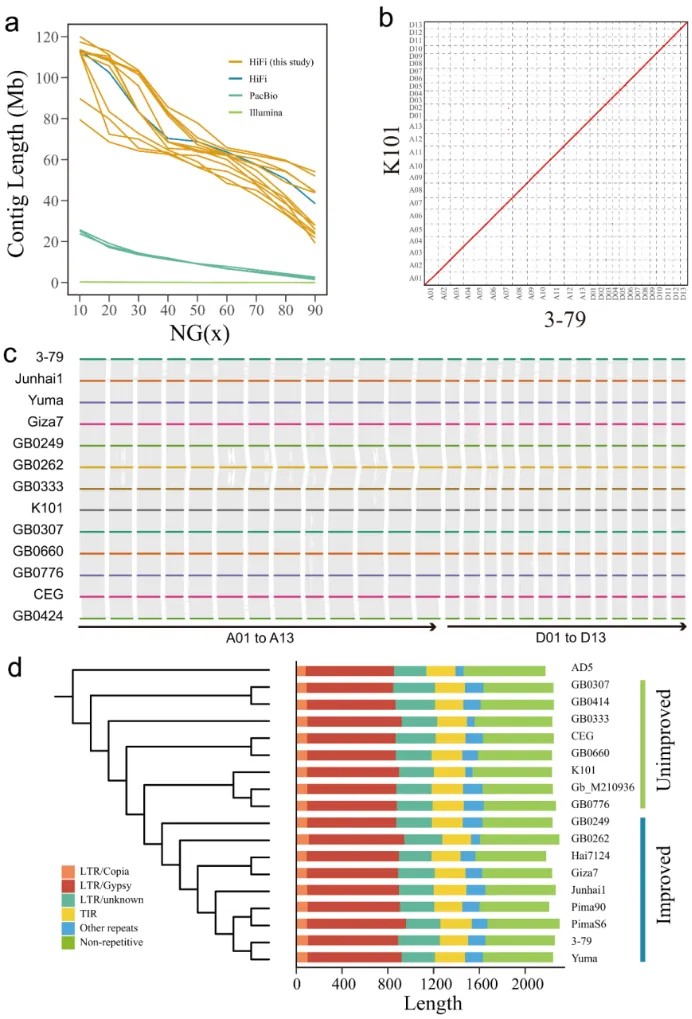 图1 从头组装12个海岛棉基因组
图1 从头组装12个海岛棉基因组
对12个从头组装和5个先前发表的海岛棉基因组开展泛基因组研究。泛基因组的尺寸大小排列组合数据显示,14个基因组就包含的99%以上的基因家族,并且基因家族数量增长曲线几乎趋于平稳,这表明这17个海岛棉材料既具有代表性,又在总体上较为完整。
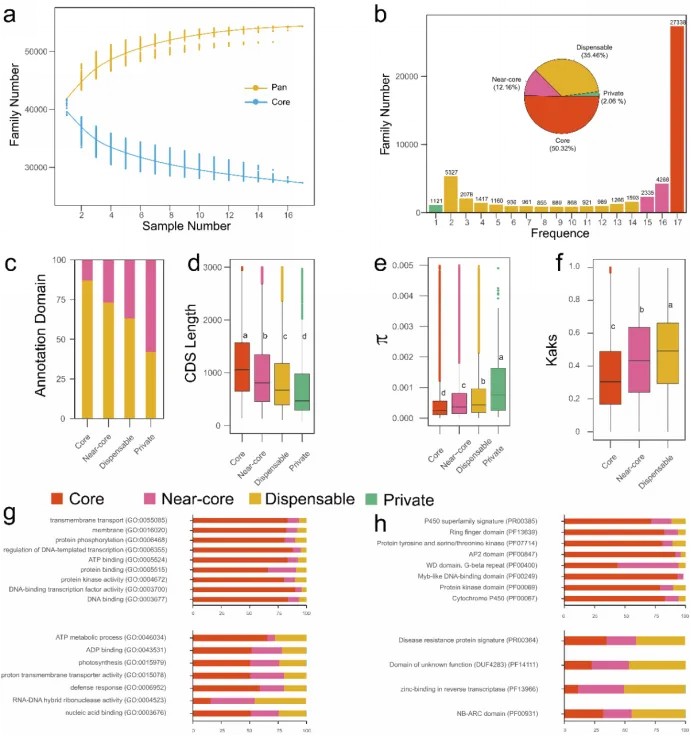 图2 海岛棉的泛基因组分析
图2 海岛棉的泛基因组分析
高质量的基因组为识别大尺寸结构变异提供遗传资源。通过比较基因组共鉴定到129,673个海岛棉的非冗余结构变异和350,995个四倍体棉花的非冗余结构变异。利用结构变异解析了海岛棉的种间渐渗,研究表明,海岛棉在加勒比海地区与陆地棉发生了广泛的种间渐渗,但仅有少量的渐渗保留到海岛棉的栽培种中,如一个位于开花基因FLK下游的结构变异起源于陆地棉,在加勒比海地区渐渗到海岛棉农家种,并遗传至了现在海岛棉栽培种,该结构变异为海岛棉在加勒比海地区通过与陆地棉种间杂交获得光周期中性性状的猜测提供了证据。
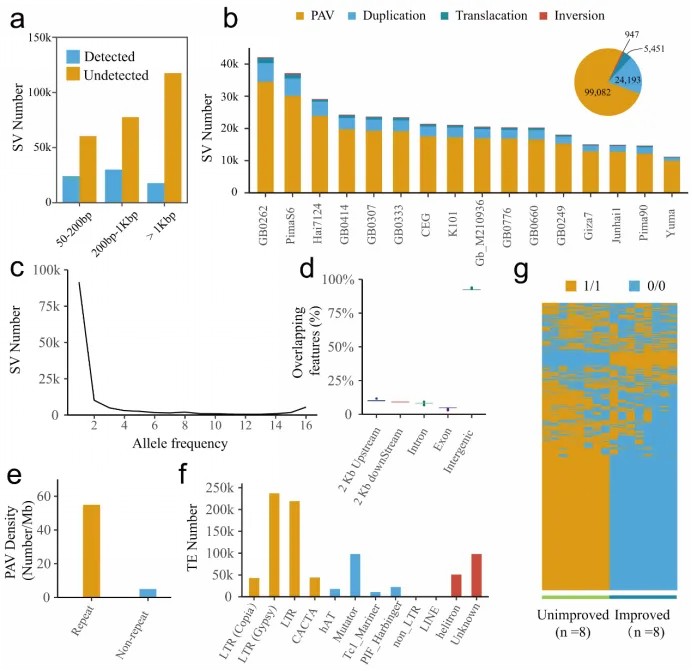 图3 海岛棉的结构变异
图3 海岛棉的结构变异
该研究通过构建图形泛基因组和开展SV-GWAS分析,鉴定到4个纤维长度,3个纤维强度和7个衣分的海岛棉遗传位点。探究这些位点优异等位变异的起源,发现三个纤维品质的有利等位变异组合(FL2/FS1+FL4/FS2+FS3)通过选择和种间渐渗结合在新疆新海棉,改良了新海棉的纤维品质;三个衣分的有利等位变异组合(LP1+LP2+LP3)通过种间杂交聚合在Pima S1,并遗传给美国皮马棉,这是皮马棉产量高的原因之一。解析海岛棉纤维品质和产量的遗传构成,发现所有衣分位点的有利等位变异均与纤维品质显著负相关,这表明海岛棉的衣分和纤维品质间存在权衡;相反三个纤维品质相关位点的优异等位变异(FL1、FL3和FL4/FS2)可在不影响海岛棉衣分的前提下进一步改良纤维品质,其中FL4/FS2已经被应用于新海棉的纤维品质改良,而FL3的优异等位变异在海岛棉群体的频率较低,尚未被充分利用。因此,未来可以将FL3的优异等位变异通过种间杂交方式整合至新海棉,实现海岛棉纤维产量和纤维品质同步改良。
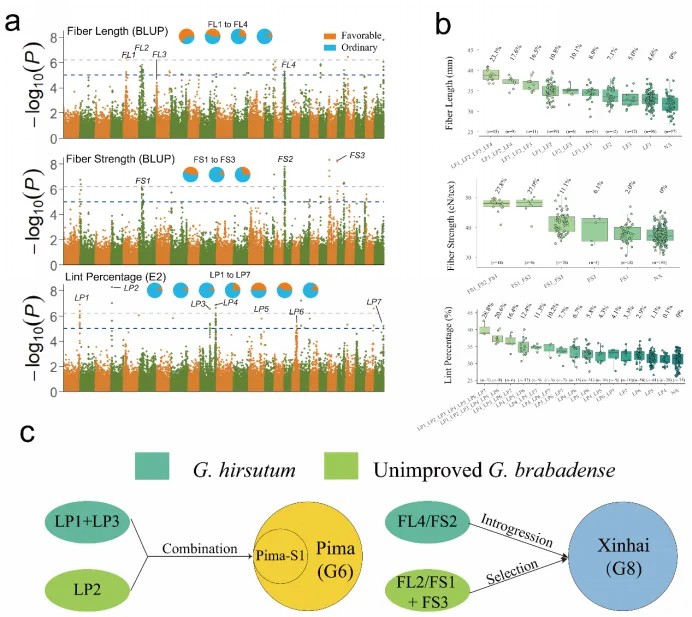
图4 SV-GWAS分析识别海岛棉纤维品质和产量的候选变异
华中农业大学作物遗传改良全国重点实验室博士生孟庆营为该论文的第一作者,袁道军副教授为通讯作者,华中农业大学棉花团队张献龙院士、金双侠教授、林忠旭教授和王茂军教授、新疆农业大学陈全家教授、新疆农业科学院棉花研究所孔杰研究员、石河子大学聂新辉教授、美国爱荷华州立大学Jonathan F. Wendel教授和美国农业部作物种质资源研究中心主任Joshua A. Udall等参与了该研究。该研究得到了国家重点研发计划、新疆自治区杰出青年基金、国家科技创新2030重大项目、华中农业大学自主创新基金和新疆人才发展基金XL等的资助。
参考文献
Yuan, D et al. The genome sequence of Sea-Island cotton (Gossypium barbadense) provides insights into the allopolyploidization and development of superior spinnable fibres. Scientific reports 5 (1), 1-16 (2015).
Wang, M et al. Reference genome sequences of two cultivated allotetraploid cottons, Gossypium hirsutum and Gossypium barbadense. Nature genetics 51 (2), 224–229 (2019).
Yuan, D. et al. Parallel and intertwining threads of domestication in allopolyploid cotton. Advance Science 8, 2003634 (2021).
 GCGI
GCGI



